近日,上海科技大学生命科学与技术学院孙博课题组与中国科学院生物与化学交叉中心刘聪课题组合作,在学术期刊《核酸研究》(Nucleic Acids Research)发表了题为“Phase-separated ParB enforces diverse DNA compaction modes and stabilizes the parS-centered partition complex”(ParB蛋白通过相分离介导多种DNA压缩模式并稳定以parS为中心的分离复合物)的研究论文,报道了染色体分离蛋白ParB通过相分离凝聚DNA、组装分离复合物的多种模式。
染色体分离是将新复制的基因组重新分配到子细胞的过程,该过程对维持遗传信息的完整性和稳定性至关重要。在大多数细菌中,染色体和低拷贝数质粒的准确分离依赖于ParABS系统,该系统由ATP酶ParA、DNA结合蛋白ParB和类着丝粒的parS DNA序列组成。其中,ParB蛋白首先结合到parS位点并向相邻的非特异性DNA扩展,随后ParB蛋白压缩DNA形成核蛋白复合物,即分离复合物。ParA酶通过水解ATP提供驱动力推动类着丝粒分离复合物向细胞两极移动。ParB-DNA核蛋白复合物的形成是染色体准确重新分配的关键。
本研究通过荧光光镊及体外蛋白相分离表征等多种技术揭示枯草芽孢杆菌的ParB(Spo0J)蛋白具备和DNA共相分离的能力,压缩DNA形成分离复合物。研究表明,除ParB与DNA的共凝聚外,预先形成的ParB凝聚体与DNA结合后能够压缩相邻的DNA,并连接远端DNA形成loop结构(图1)。此外,CTP促进了低浓度ParB在parS位点形成凝聚体,以parS为中心的ParB-DNA共同凝聚体能够承受数十皮牛破坏力。这些发现表明ParB通过多聚实现高阶复合物的组装并维持其稳定性。总体而言,该工作报道了ParB通过相分离介导的多种DNA压缩模式,为细菌分离复合物的动态组装和维护提供了新见解,也为理解ParABS系统介导的染色体分离提供了重要的分子基础。
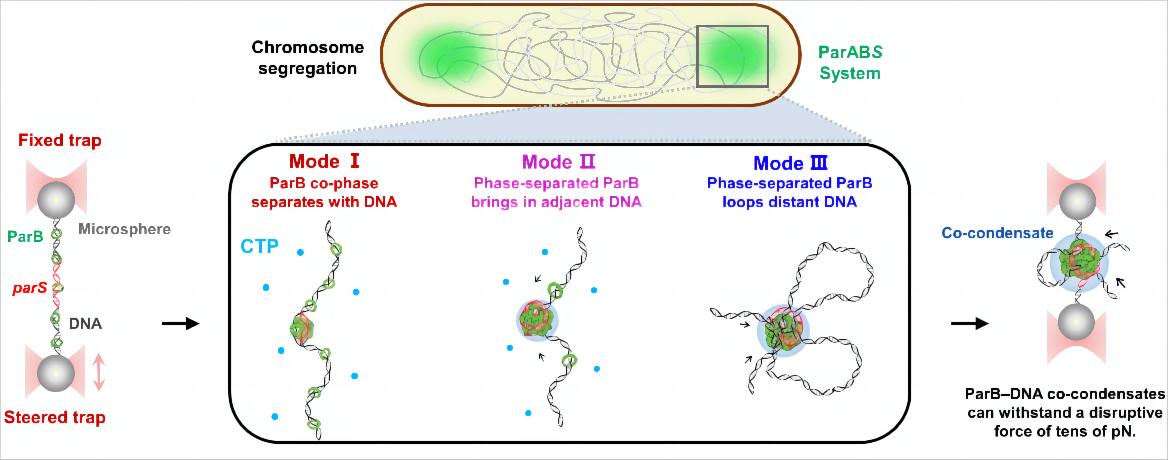
图1:ParB蛋白通过相分离驱动分离复合物组装的多种模式。
该论文中,上海科技大学生命科学与技术学院博士生赵艺琳、郭丽娟(已毕业)及中国科学院生物与化学交叉中心博士后胡姣姣为共同第一作者,孙博教授和刘聪研究员为共同通讯作者,上海科技大学为第一完成单位。


 沪公网安备 31011502006855号
沪公网安备 31011502006855号