近日,上海科技大学生命科学与技术学院研究团队利用核磁共振技术,首次解析了鼠源RIPK1(mRIPK1)蛋白RHIM结构域淀粉样纤维的三维结构,并在此基础上表征了mRIPK1/mRIPK3复合物纤维结构,及两种蛋白在形成异质纤维过程中的结构变化。该成果发表在《自然-通讯》(Nature Communications),论文题为“The structure of mouse RIPK1 RHIM-containing domain as a homo-amyloid and in RIPK1/RIPK3 complex”。

细胞凋亡(apoptosis)和细胞程序性坏死(necroptosis)是常见的两种程序性细胞死亡形式,其中受体相互作用蛋白激酶1(RIPK1)和受体相互作用蛋白激酶3(RIPK3)在程序性细胞死亡信号通路中起关键作用。通过RIPK1和RIPK3,程序性细胞死亡参与了很多组织损伤、炎症和神经系统疾病的发生和发展。RIPK1是多种信号通路的重要上游调节因子,包括炎症、细胞凋亡和程序性坏死。RIPK1由N端的激酶结构域(KD)、中间结构域(ID)以及C端的死亡结构域(DD)组成。中间结构域是一个超过250个残基的序列,包含一个约20个残基的RIP同型相互作用基序(RHIM)。RHIM结构域参与淀粉样纤维的形成并通过与其他含RHIM的蛋白相互作用,如RIPK3,形成具有淀粉样结构特性的蛋白复合物,称为坏死小体。
研究团队通过生物固态核磁等技术解析了mRIPK1的RHIM结构域形成的淀粉样纤维三维结构,它由4段β-strand(β折叠链)堆叠形成“N”型的结构。研究发现mRIPK1和人源RIPK3(hRIPK3)形成的淀粉样纤维结构非常相似,但与鼠源RIPK3(mRIKPK3)的结构存在部分差异,尽管RHIM蛋白结构域之间具有保守性。
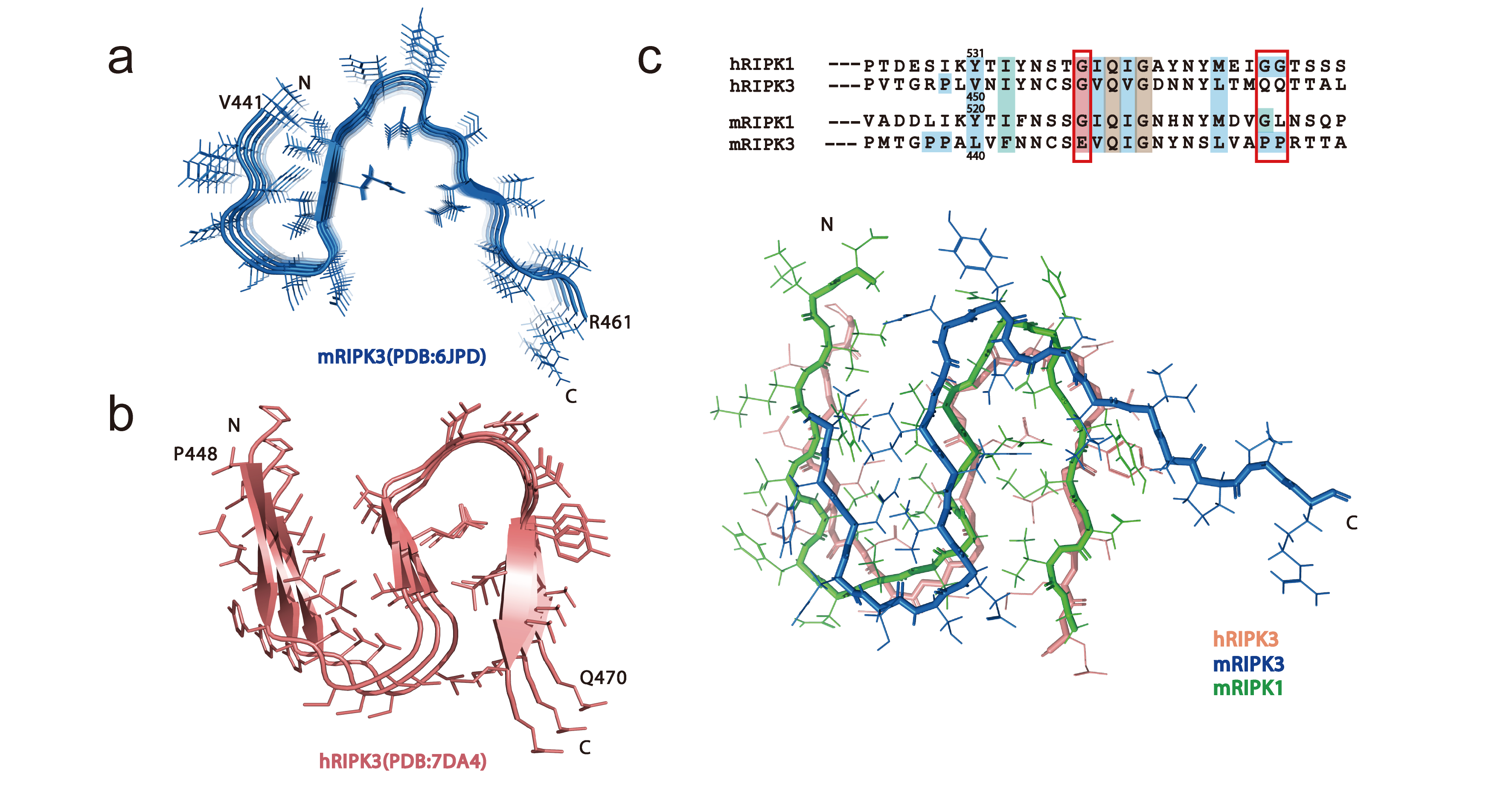
图1.mRIPK1与hRIPK3、mRIPK3纤维结构的比较
进一步比较发现,mRIPK3的β3与中间的β2靠近程度不如其他两个纤维结构(mRIPK1和hRIPK3)。通过氨基酸序列分析,发现mRIPK3的核心四联体( I/VQI/VG)前面是E447,不同于其他两个(都为G)(图1)。这可能是由于残基E是带电荷的氨基酸,并有长的侧链,不利于参与β-strand间的相互作用。
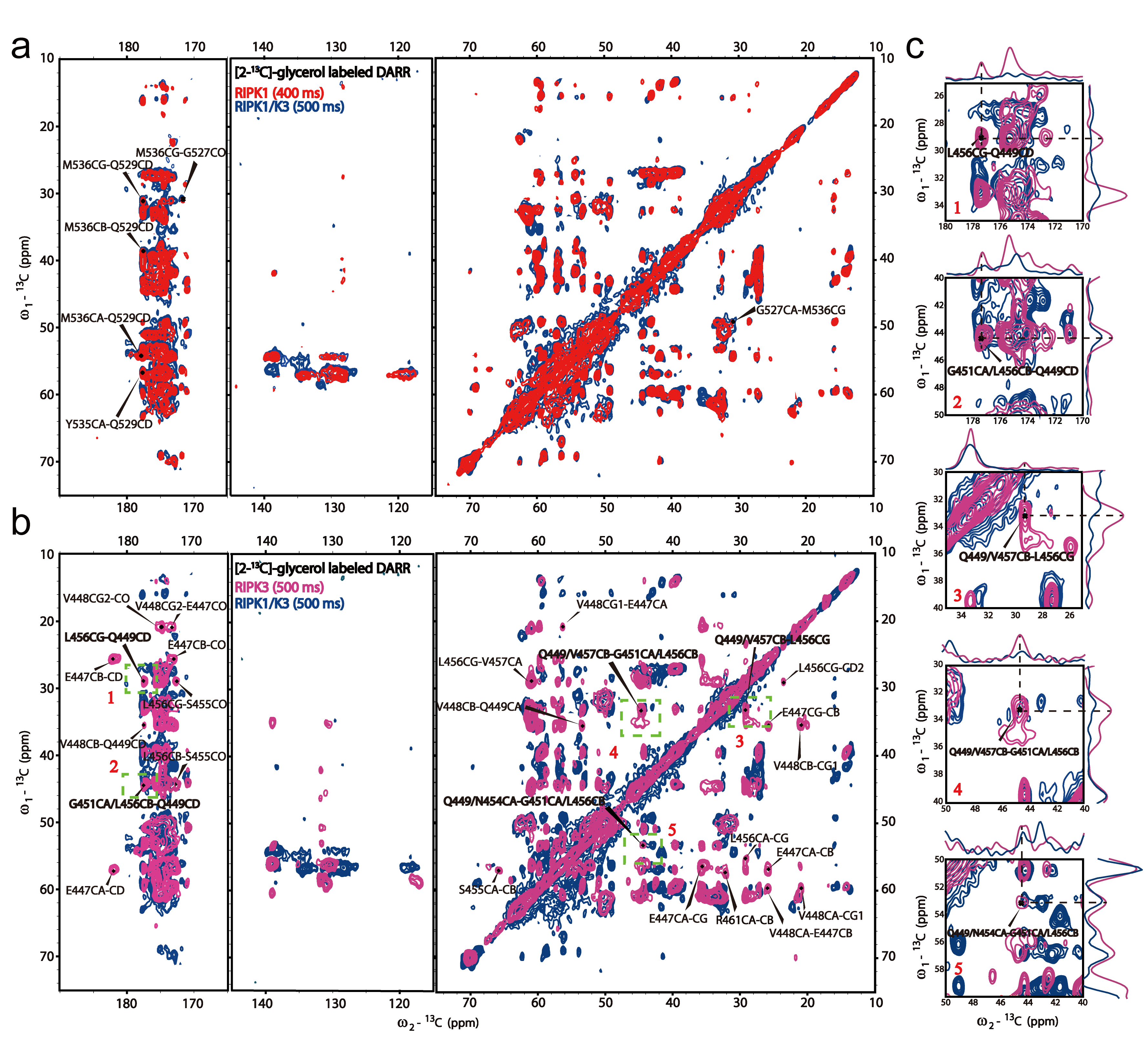
图2.[2-13C]-甘油标记的异源复合物纤维与同源纤维的二维DARR核磁图谱比较
研究还发现,mRIPK3与RIPK1相互作用后,mRIPK1结构几乎无明显变化,而mRIPK3却表现出明显的交叉峰强度的降低。与同质mRIPK3纤维结构相比,异质纤维复合物mRIPK3中定义β3链和β2链相互作用的交叉峰在谱图上消失(图2),说明异质mRIPK3最后一条β-strand远离了中心β-strand,使得mRIPK3结构动态性增加,这有助于进一步进行mRIPK3单体招募和交换。
本研究为了解不同蛋白与RHIM结构域之间的相互作用提供了重要信息,这将有助于深入理解细胞坏死的调控机制。
上海科技大学生命学院2021级博士研究生刘静为该论文的第一作者,上科大生命学院生物物理技术平台主任王健和武汉科技大学教授陆珺霞为共同通讯作者。上海科技大学为第一完成单位。


 沪公网安备 31011502006855号
沪公网安备 31011502006855号