代谢状态改变是癌症细胞的典型特征之一。代谢依赖性靶点通过靶向癌症特征性代谢状态变化实现对癌细胞的选择性杀伤抑制,系统发掘癌症细胞的代谢依赖性靶点目前依然是一个未解决的重要问题。

近日,上海科技大学生命科学与技术学院刘雪松课题组在国际学术期刊《细胞报告》(Cell Reports)上发表了题为“Precise metabolic dependencies of cancer through deep learning and validations”的研究论文,构建了基于图深度学习的癌症代谢依赖性预测模型“DeepMeta”,该模型依据转录组和代谢网络信息,首次实现癌症样本代谢依赖性靶点的精确预测(图1)。
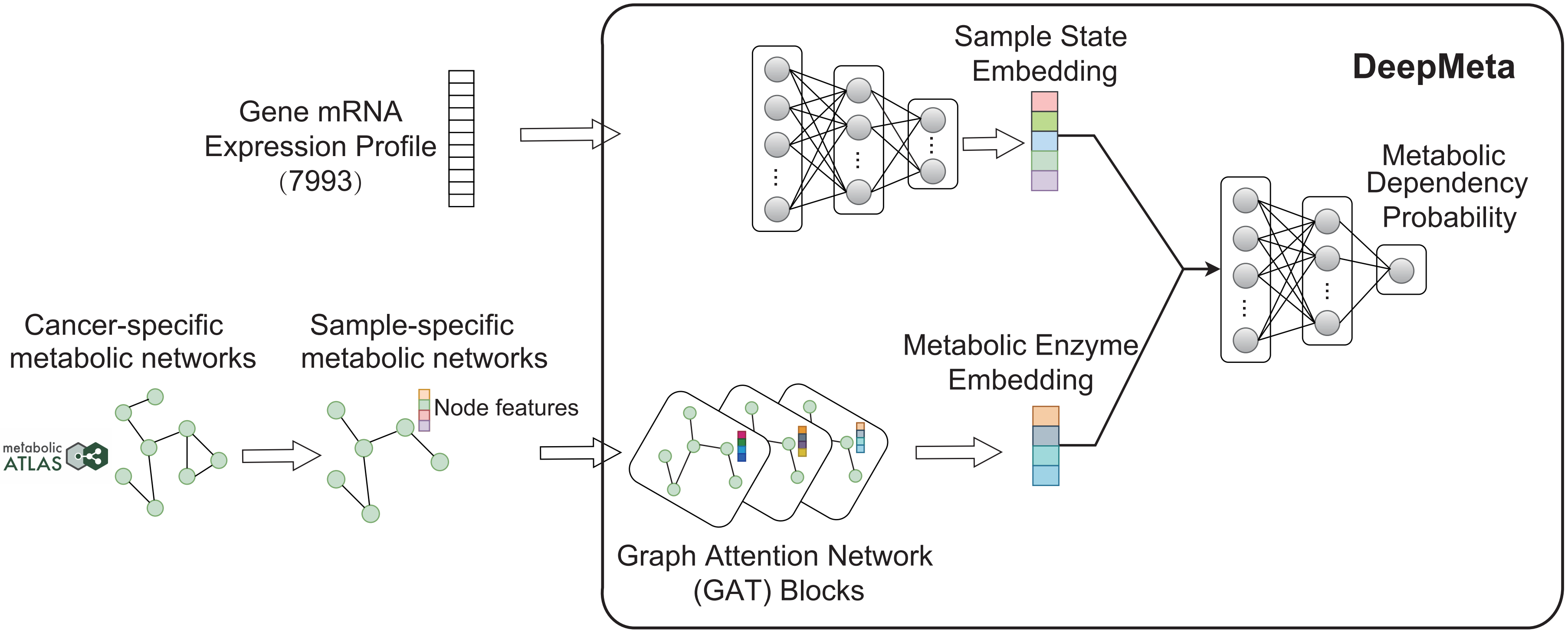
图1.癌症代谢依赖性预测模型——DeepMeta
抗代谢药物是一类临床上广泛应用的癌症化疗药物,目前还没有很好的标志物用于其临床治疗效果预测。本研究应用DeepMeta预测了TCGA癌症样本的依赖性代谢通路,发现DeepMeta预测的嘧啶代谢通路依赖的癌症患者对抗嘧啶代谢药物(卡培他滨、培美曲塞、吉西他滨、氟尿嘧啶)有着显著的治疗效果,这不仅证明了DeepMeta可靠预测性能,也为抗代谢药物的临床应用提供了新的预测标志物(图2)。
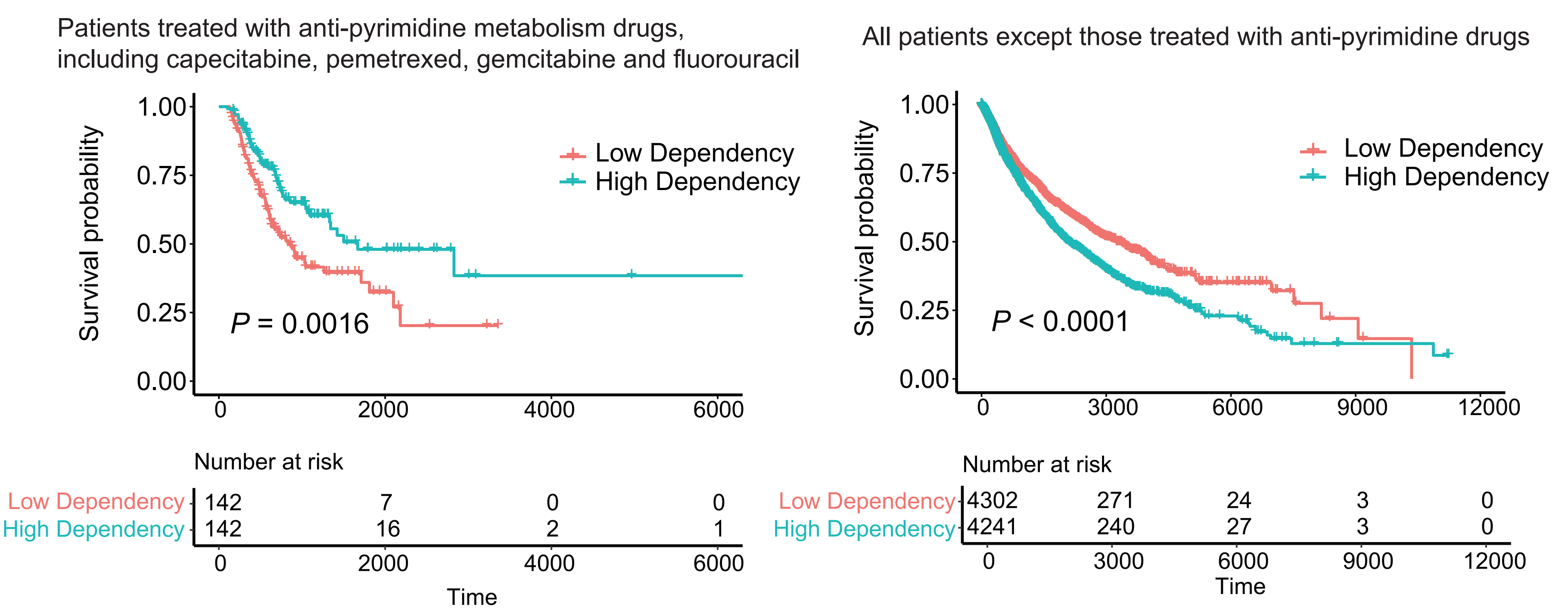
图2.DeepMeta预测的嘧啶代谢通路依赖的癌症对抗嘧啶代谢药物有着显著的治疗效果
研究人员进一步应用DeepMeta模型预测了常见不可成药的癌症驱动基因(包括TP53、CTNNB1和MYC)变异的代谢依赖性,并进行了湿实验验证。实验发现CTNNB1激活突变(T41A)导致对嘧啶和嘌呤代谢的依赖,通过药物抑制嘧啶和嘌呤代谢可以选择性地抑制携带CTNNB1 T41A突变细胞的增殖(图3)。
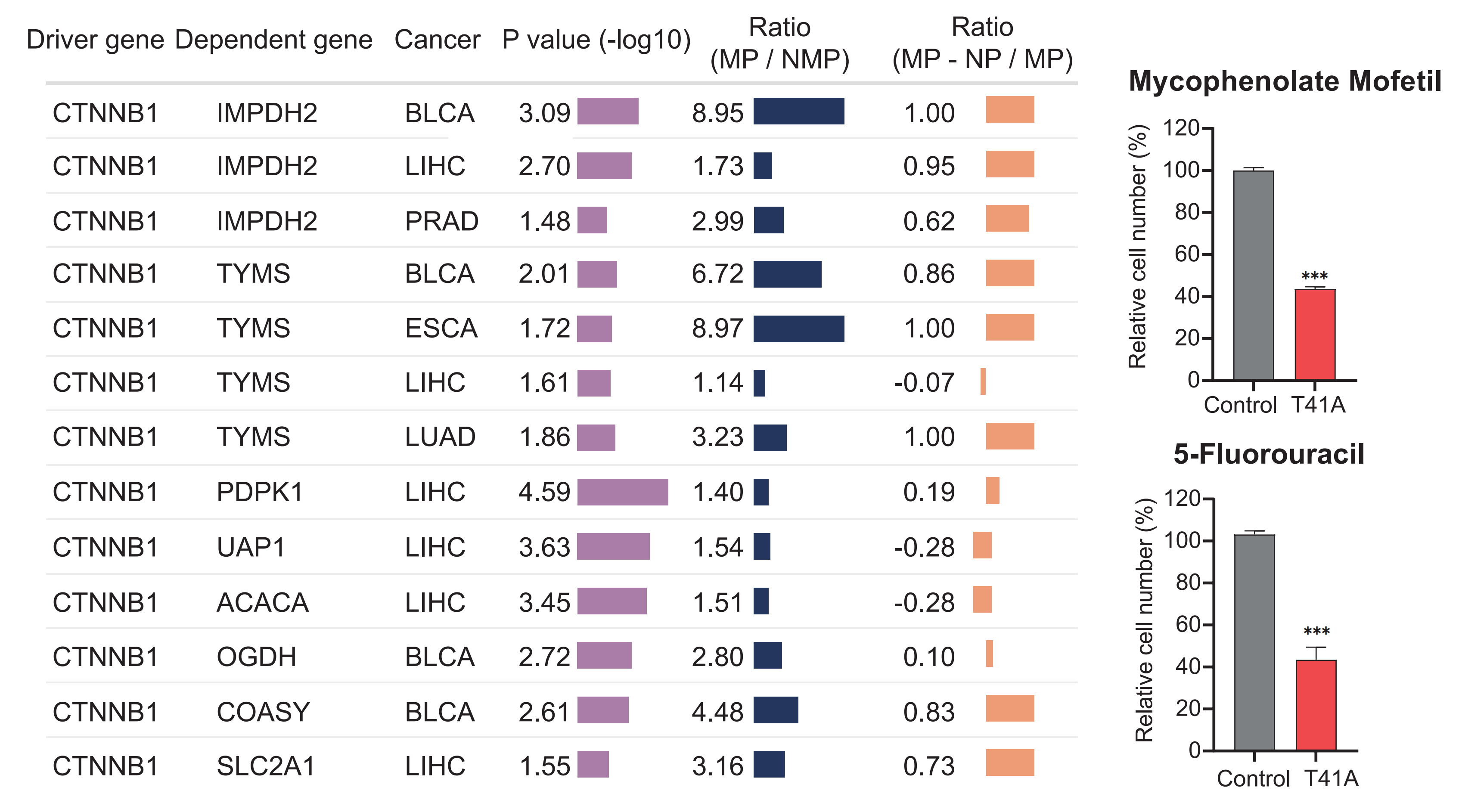
图3. CTNNB1激活突变(T41A)导致对嘧啶和嘌呤代谢的依赖
本研究为系统挖掘癌症代谢依赖性靶点提供了新的工具,为不可成药的癌症驱动变异提供了新的靶向治疗策略,对癌症精准诊断治疗临床实践具有潜在的应用价值。
上海科技大学生命学院2024届博士毕业生吴涛、赵翔宇和2025届硕士毕业生张钰为本文共同第一作者。生命学院刘雪松教授为本文通讯作者。上海科技大学为第一完成单位和通讯单位。
论文链接:https://www.cell.com/cell-reports/fulltext/S2211-1247(25)00716-8


 沪公网安备 31011502006855号
沪公网安备 31011502006855号